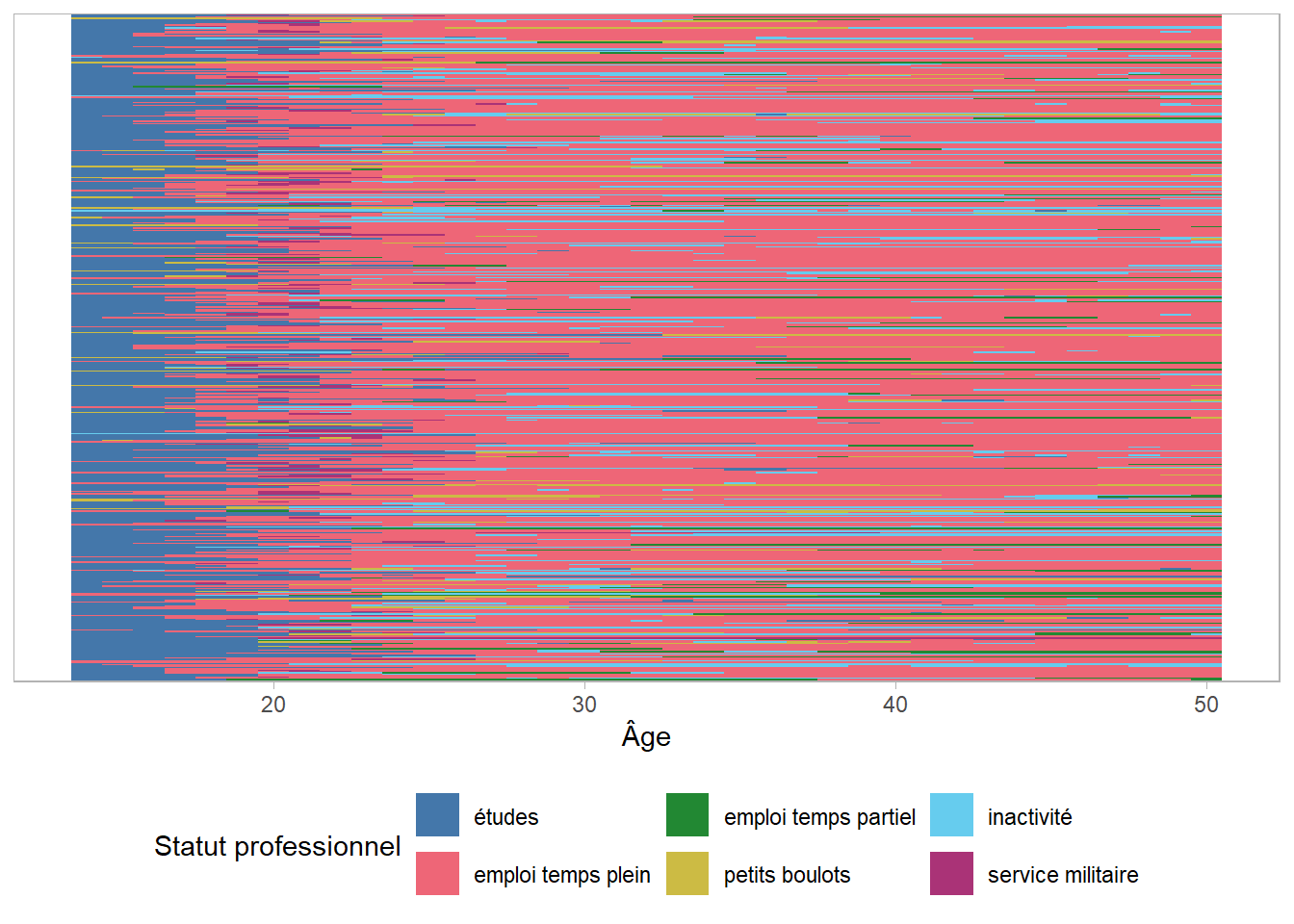
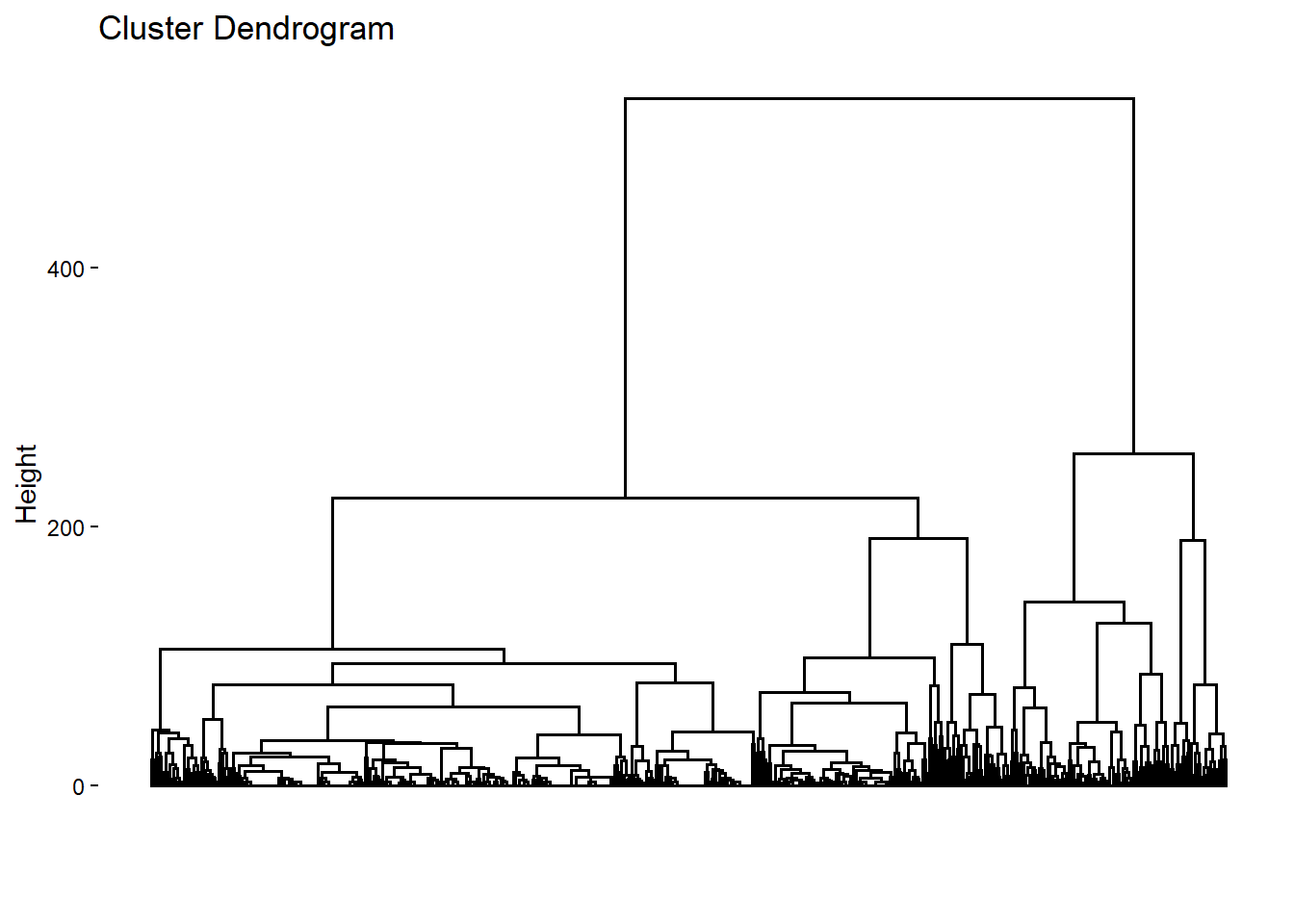
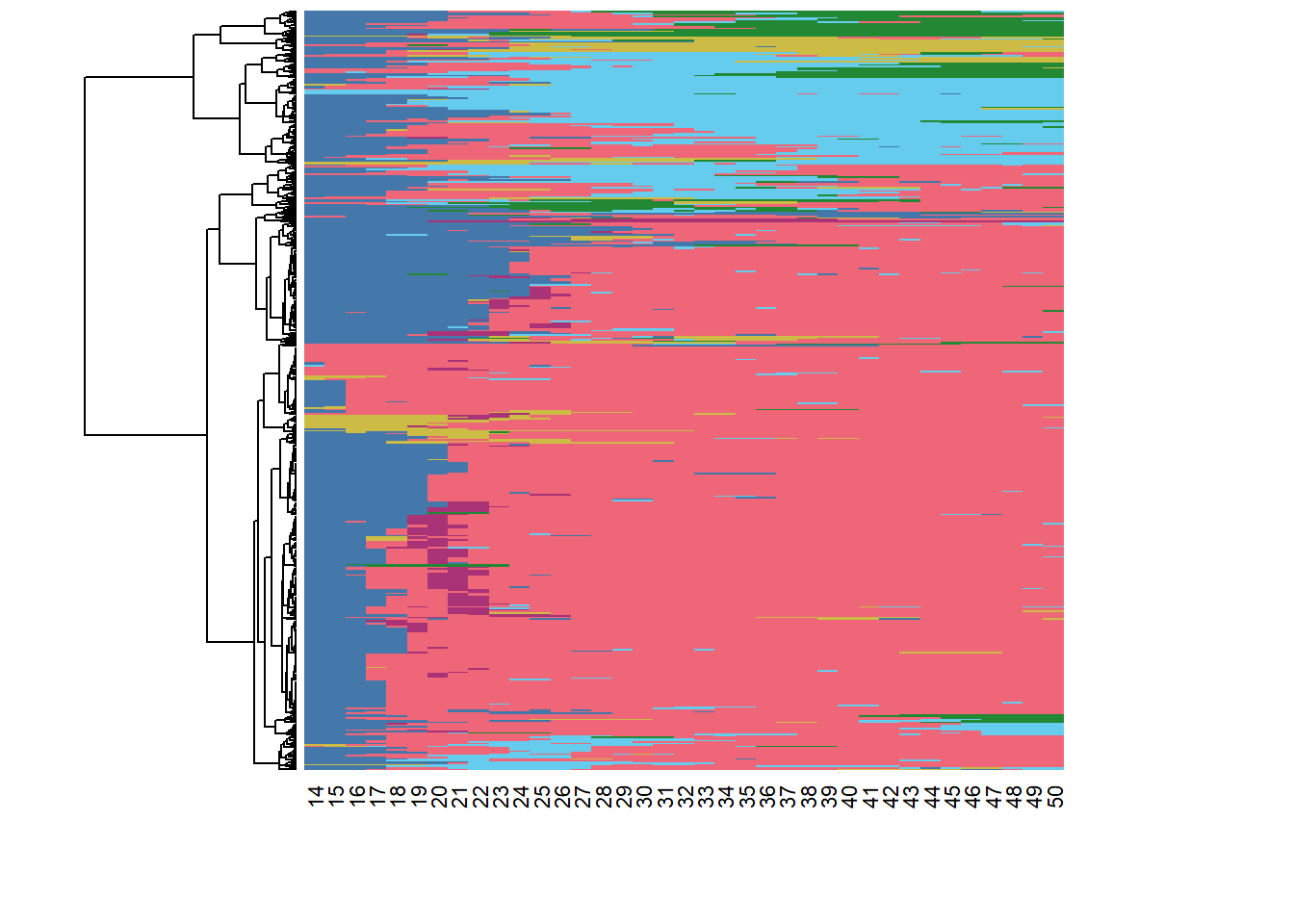
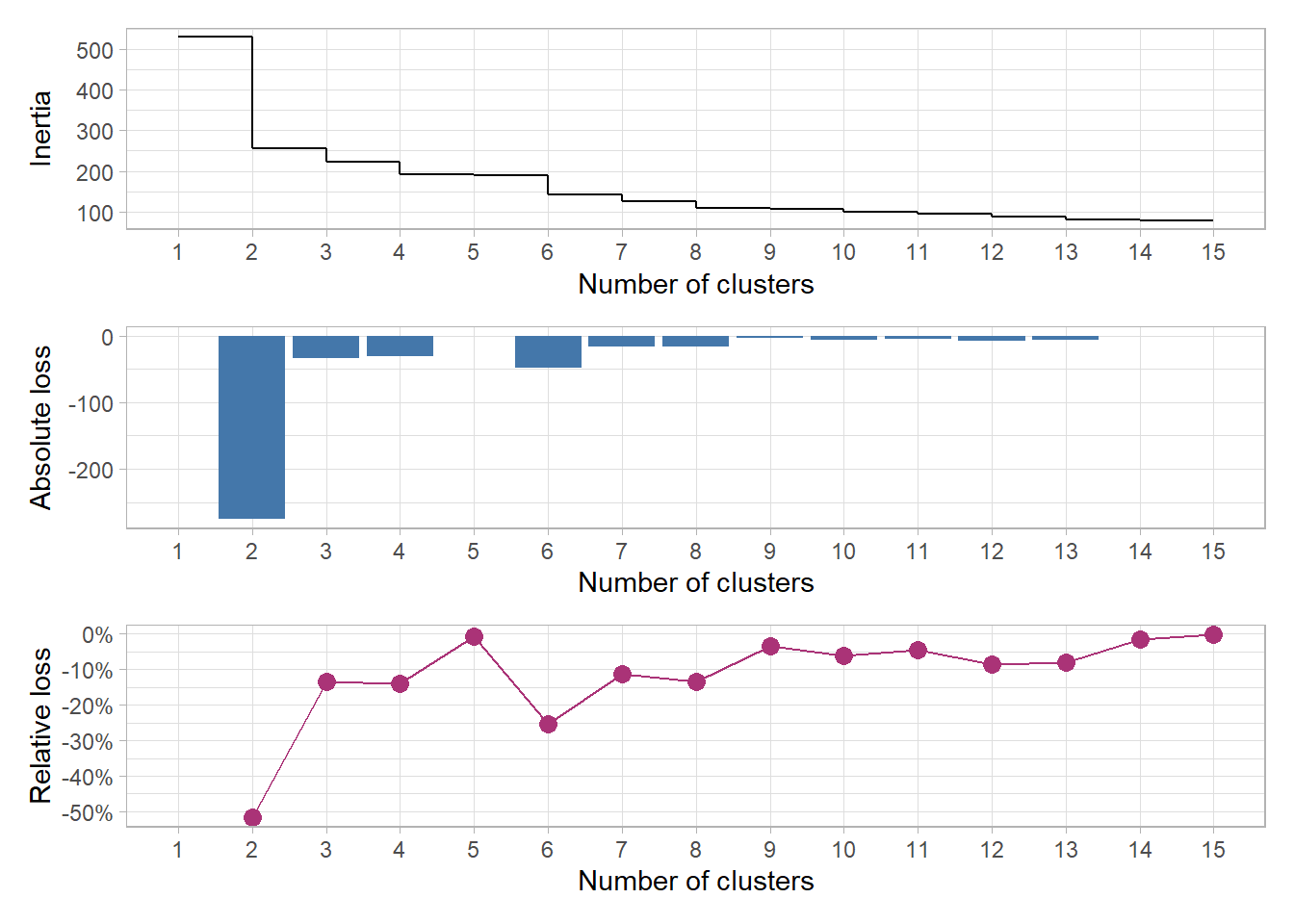
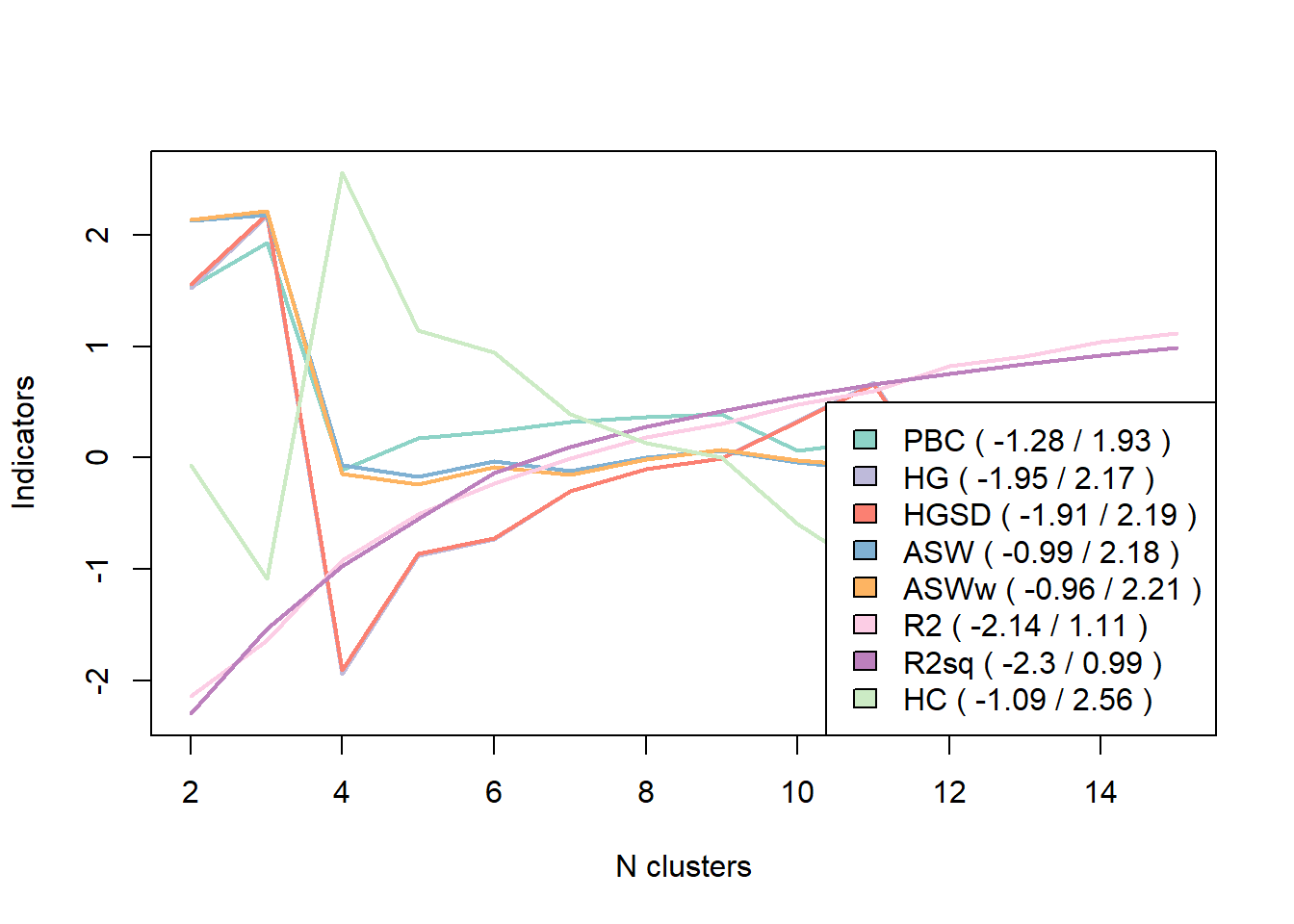
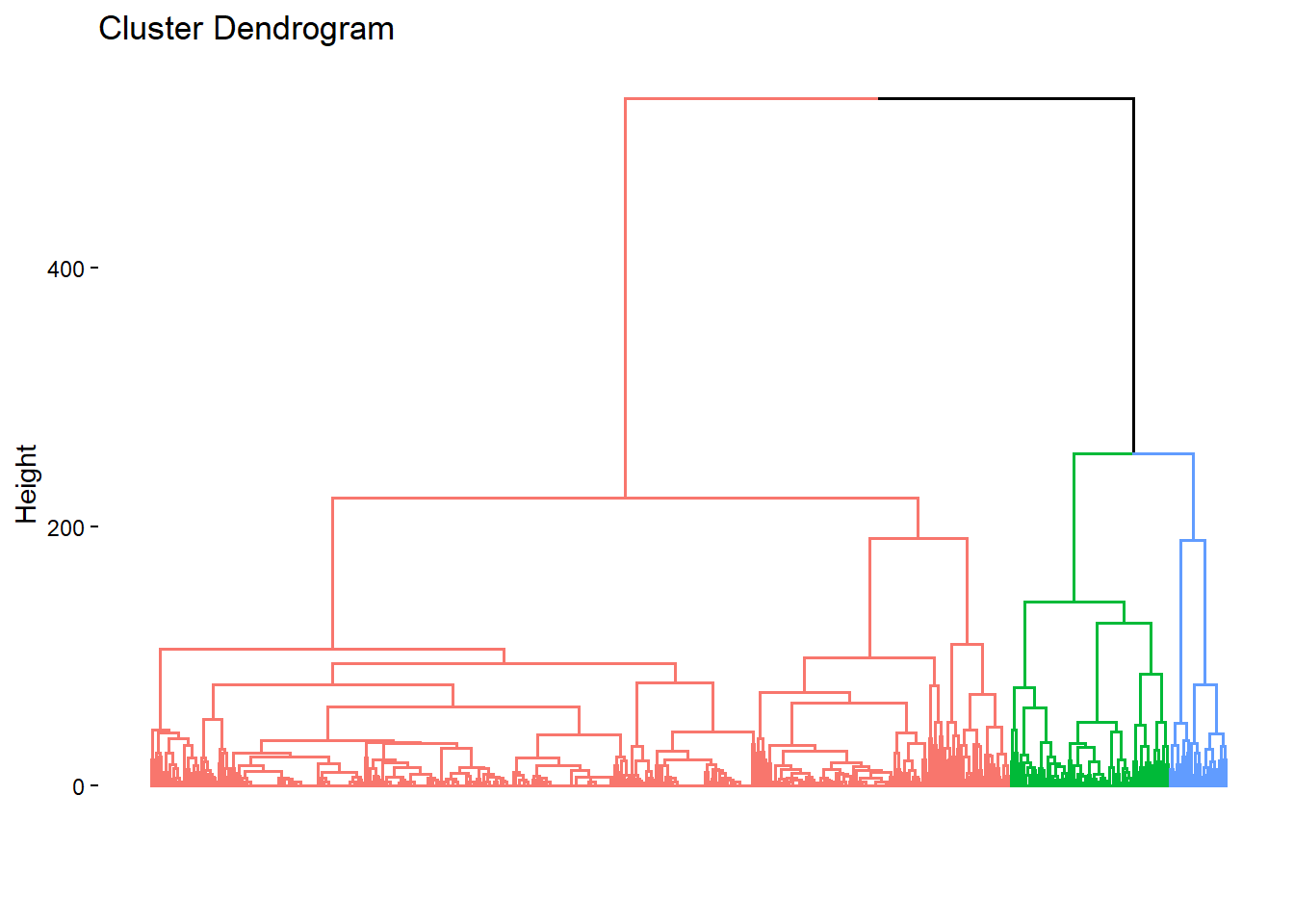
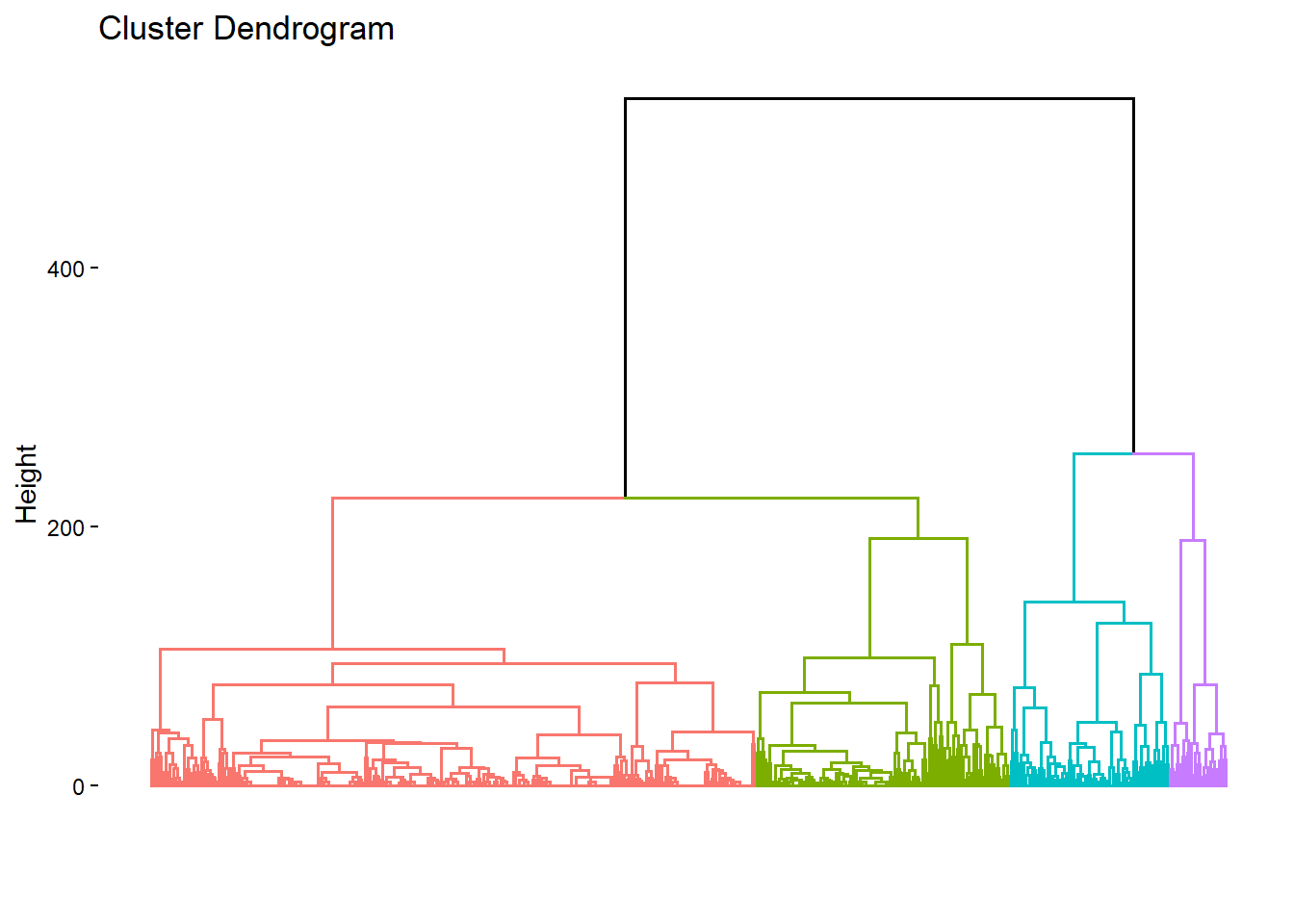
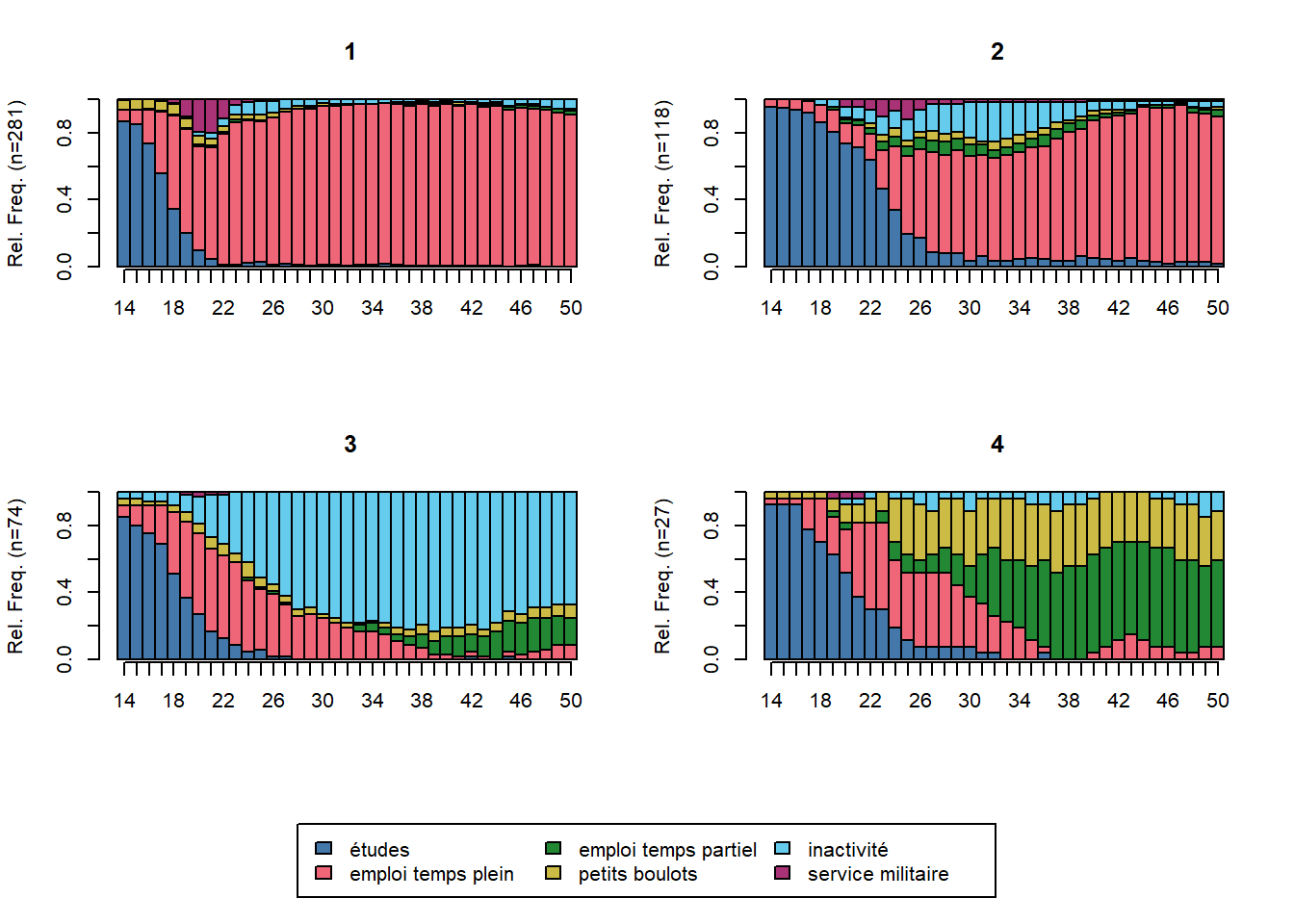
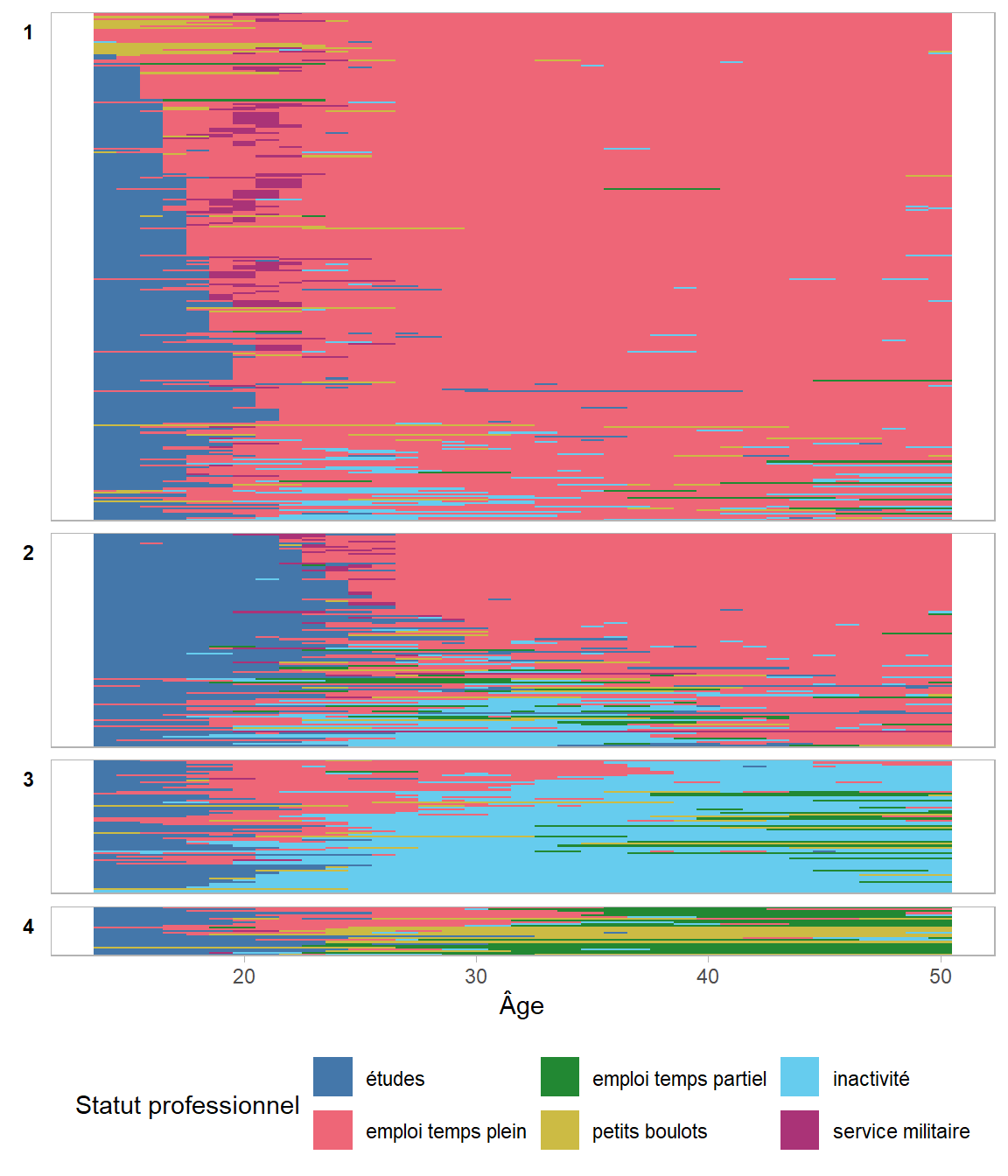
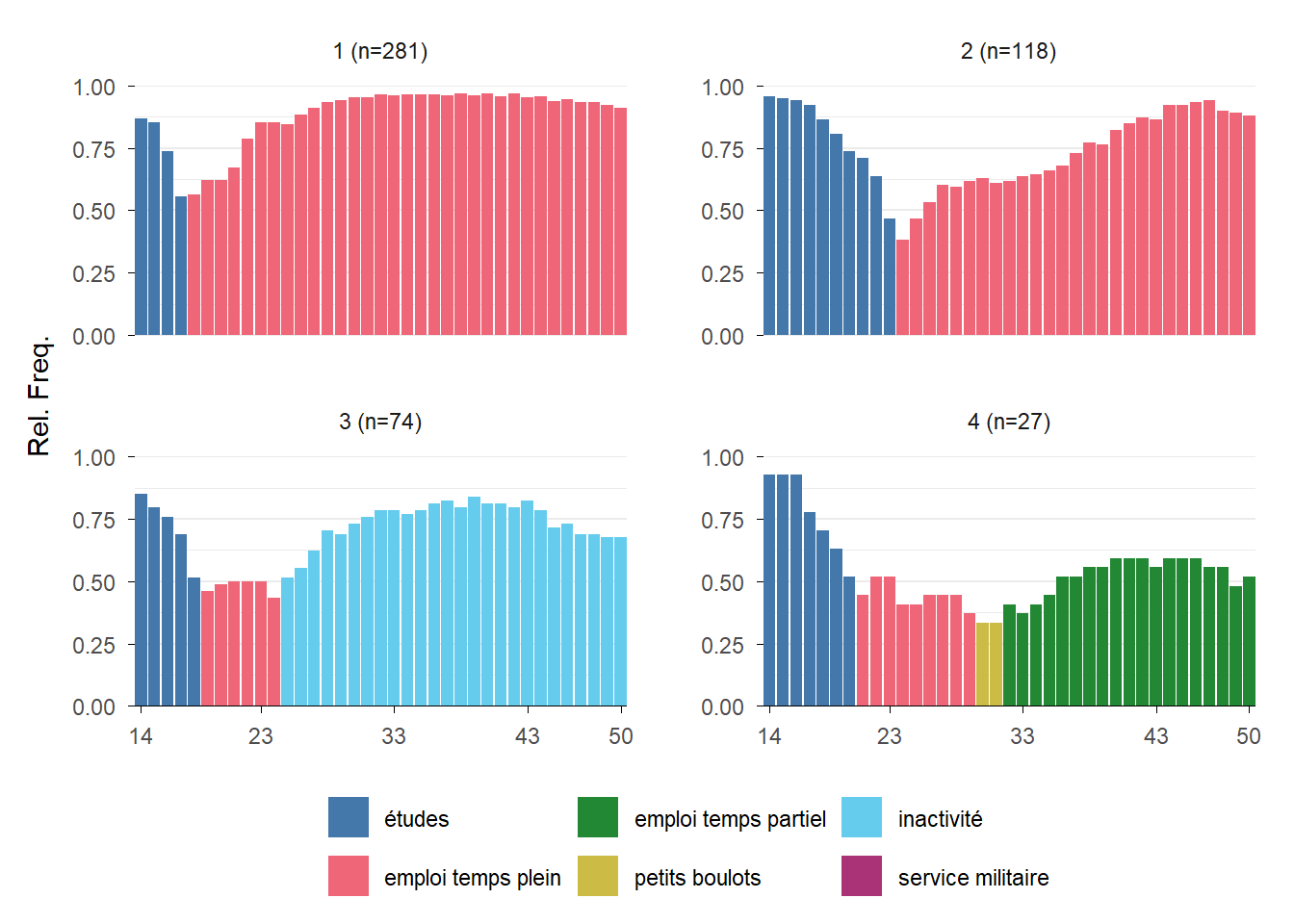
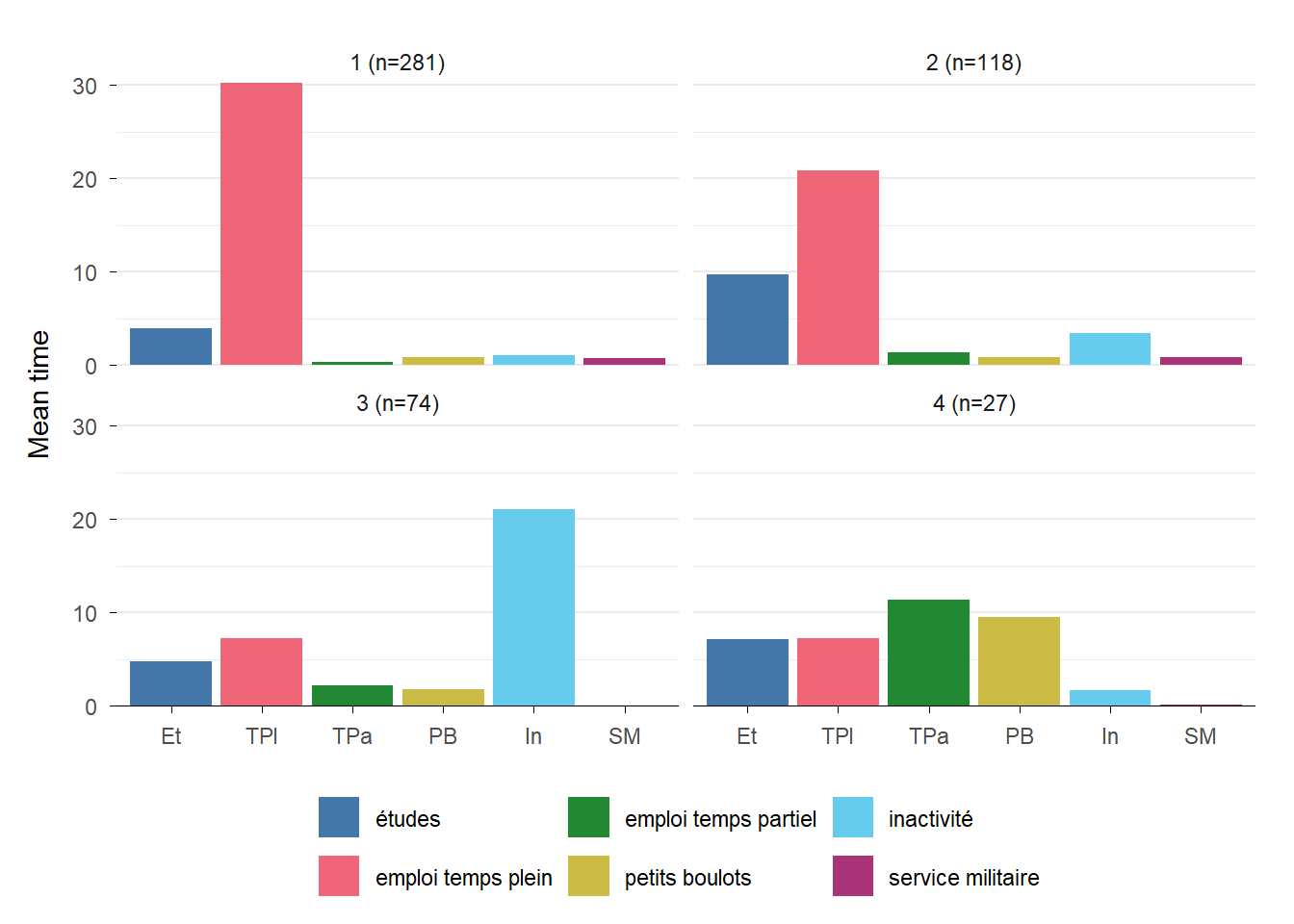
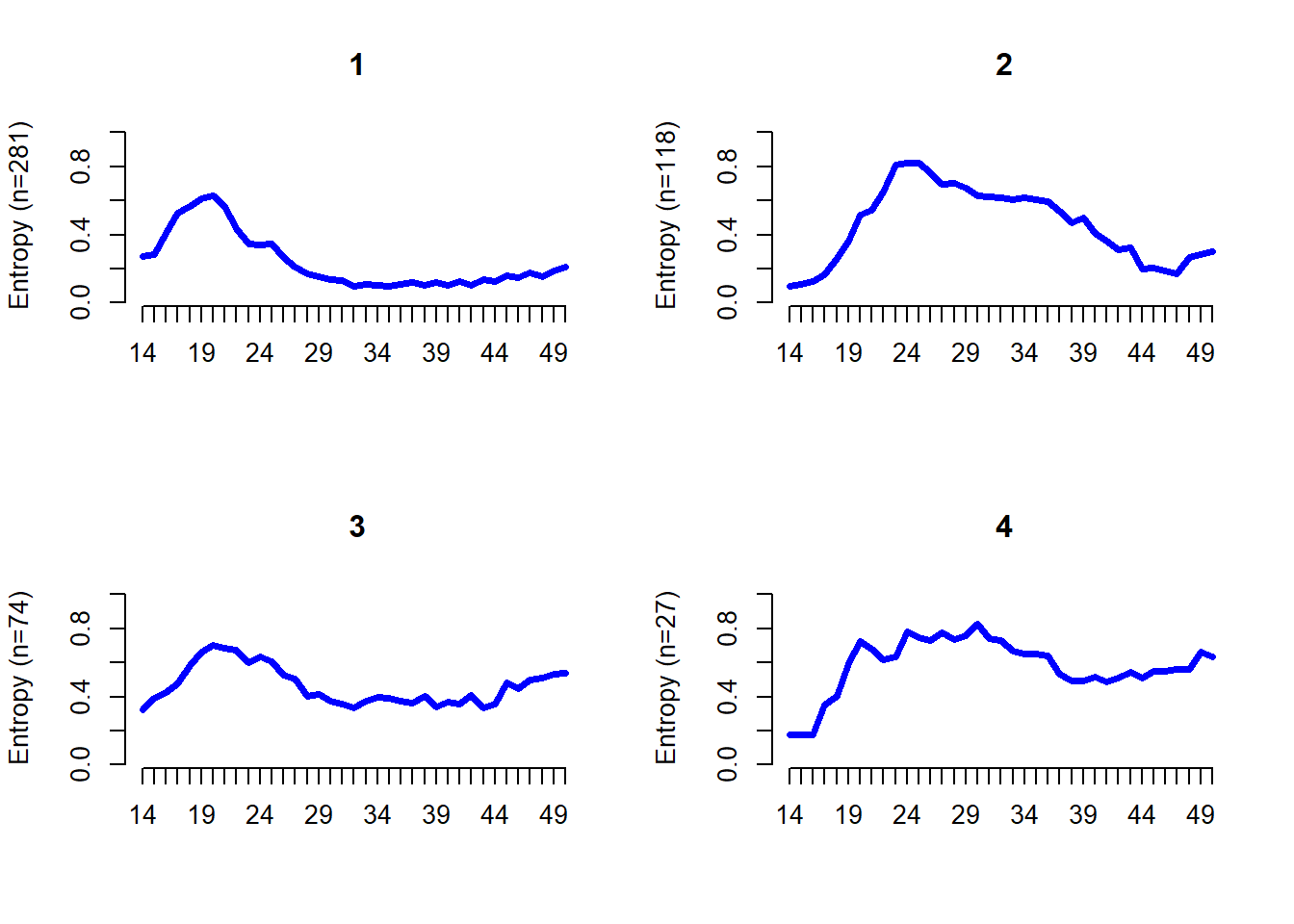
Rows: 500
Columns: 37
$ sact14 <dbl> 2, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 1, 1, 1, 1, 1, …
$ sact15 <dbl> 2, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 1, 2, 2, 1, 1, 1, 1, 1, …
$ sact16 <dbl> 2, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 2, 2, 1, 2, 2, 1, 1, 1, 1, 1, …
$ sact17 <dbl> 2, 1, 1, 1, 1, 1, 2, 2, 2, 2, 1, 2, 2, 1, 2, 2, 1, 2, 1, 1, 1, …
$ sact18 <dbl> 2, 1, 2, 2, 1, 1, 2, 2, 2, 2, 1, 2, 2, 1, 2, 2, 1, 2, 1, 1, 1, …
$ sact19 <dbl> 2, 3, 2, 2, 1, 1, 2, 2, 2, 2, 1, 2, 2, 1, 2, 2, 2, 2, 1, 1, 1, …
$ sact20 <dbl> 2, 3, 2, 6, 2, 2, 5, 2, 6, 2, 1, 2, 2, 1, 2, 2, 2, 2, 1, 1, 1, …
$ sact21 <dbl> 2, 3, 2, 2, 2, 2, 5, 6, 6, 2, 1, 2, 5, 5, 2, 2, 2, 2, 1, 1, 1, …
$ sact22 <dbl> 2, 3, 2, 2, 2, 2, 5, 6, 2, 2, 1, 2, 5, 5, 2, 2, 2, 2, 1, 1, 1, …
$ sact23 <dbl> 2, 3, 2, 2, 2, 2, 5, 2, 2, 2, 1, 2, 5, 5, 2, 4, 6, 2, 1, 1, 2, …
$ sact24 <dbl> 2, 3, 2, 2, 2, 5, 3, 2, 2, 2, 1, 2, 5, 5, 2, 4, 6, 2, 5, 1, 2, …
$ sact25 <dbl> 2, 3, 2, 2, 2, 5, 3, 2, 2, 2, 2, 2, 5, 5, 2, 4, 6, 2, 5, 1, 2, …
$ sact26 <dbl> 2, 3, 2, 2, 2, 5, 3, 2, 2, 2, 2, 2, 5, 5, 2, 4, 6, 2, 5, 1, 2, …
$ sact27 <dbl> 2, 3, 2, 2, 2, 2, 3, 2, 2, 2, 2, 2, 5, 5, 2, 4, 2, 2, 5, 2, 2, …
$ sact28 <dbl> 2, 3, 5, 2, 2, 2, 3, 2, 2, 2, 2, 5, 5, 5, 2, 4, 2, 2, 5, 2, 2, …
$ sact29 <dbl> 2, 3, 2, 2, 2, 2, 3, 2, 2, 2, 2, 5, 5, 5, 2, 4, 2, 2, 5, 2, 2, …
$ sact30 <dbl> 2, 3, 5, 2, 2, 2, 3, 2, 2, 2, 2, 5, 5, 5, 2, 4, 2, 2, 5, 2, 2, …
$ sact31 <dbl> 2, 3, 5, 2, 2, 2, 3, 2, 2, 2, 2, 5, 5, 5, 2, 4, 2, 2, 5, 2, 5, …
$ sact32 <dbl> 2, 3, 5, 2, 2, 2, 5, 2, 2, 2, 2, 5, 5, 5, 2, 4, 2, 2, 5, 2, 5, …
$ sact33 <dbl> 2, 3, 5, 2, 2, 2, 5, 2, 2, 2, 2, 5, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact34 <dbl> 2, 3, 5, 2, 2, 2, 5, 2, 2, 2, 2, 5, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact35 <dbl> 2, 3, 5, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact36 <dbl> 2, 3, 5, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact37 <dbl> 2, 3, 5, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact38 <dbl> 2, 2, 5, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact39 <dbl> 2, 2, 5, 2, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact40 <dbl> 2, 2, 5, 2, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact41 <dbl> 2, 2, 5, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact42 <dbl> 2, 2, 5, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 5, 2, 2, 3, 2, 5, …
$ sact43 <dbl> 2, 2, 5, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 2, 2, 2, 3, 2, 5, …
$ sact44 <dbl> 2, 2, 5, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 2, 2, 2, 3, 2, 5, …
$ sact45 <dbl> 2, 2, 3, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 2, 2, 2, 3, 2, 5, …
$ sact46 <dbl> 2, 2, 3, 2, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 2, 2, 2, 2, 3, 2, 5, …
$ sact47 <dbl> 2, 2, 3, 2, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 2, 2, 2, 2, 3, 2, 5, …
$ sact48 <dbl> 2, 2, 3, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 2, 2, 2, 3, 2, 5, …
$ sact49 <dbl> 2, 2, 3, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 2, 2, 2, 3, 2, 5, …
$ sact50 <dbl> 2, 3, 3, 2, 2, 2, 2, 2, 2, 2, 2, 5, 5, 5, 2, 2, 2, 2, 3, 2, 5, …