Analyse de survie
Ce chapitre est évoqué dans le webin-R #15 (analyse de survie) sur YouTube.
Ce chapitre est évoqué dans le webin-R #18 (trajectoires de soins : un exemple de données longitudinales 2 : analyse de survie) sur YouTube.
Ce chapitre est évoqué dans le webin-R #21 (trajectoires de soins : un exemple de données longitudinales 5 : modèle à observations répétée, régression logistique ordinale GEE & analyse de survie multi-états) sur YouTube.
Ressources en ligne
L’extension centrale pour l’analyse de survie est survival.
Un très bon tutoriel (en anglais et en 3 étapes), introduisant les concepts de l’analyse de survie, des courbes de Kaplan-Meier et des modèles de Cox et leur mise en oeuvre pratique sous R est disponible en ligne :
- http://www.sthda.com/english/wiki/survival-analysis-basics
- http://www.sthda.com/english/wiki/cox-proportional-hazards-model
- http://www.sthda.com/english/wiki/cox-model-assumptions
Pour un autre exemple (toujours en anglais) d’analyse de survie avec survival, on pourra se référer à https://rpubs.com/vinubalan/hrsurvival.
Pour représenter vos résultats avec ggplot2, on pourra avoir recours à l’extension survminer présentée en détails sur son site officiel (en anglais) : http://www.sthda.com/english/rpkgs/survminer/. On pourra également avoir recours à la fonction ggsurv de l’extension GGally présentée à l’adresse http://ggobi.github.io/ggally/#ggallyggsurv.
A noter, il est possible d’utiliser la fonction step sur un modèle de Cox, pour une sélection pas à pas d’un meilleur modèle basé sur une minimisation de l’AIC (voir le chapitre sur la régression logistique).
L’excellente extension broom peut également être utilisée sur des modèles de survie (Kaplan-Meier ou Cox) pour en convertir les résultats sous la forme d’un tableau de données.
Pour approfondir les possibilités offertes par l’extension survival, on pourra également consulter les différentes vignettes fournies avec l’extension (voir https://cran.r-project.org/package=survival).
Un exemple concret : mortalité infanto-juvénile
Dans cet exemple, nous allons utiliser le jeu de données fecondite fourni par l’extension questionr. Ce jeu de données comporte trois tableaux de données : menages, femmes et enfants.
Nous souhaitons étudier ici la survie des enfants entre la naissance et l’âge de 5 ans. Dans un premier temps, nous comparerons la survie des jeunes filles et des jeunes garçons. Dans un second temps, nous procéderons à une analyse multivariée en prenant en compte les variables suivantes :
- sexe de l’enfant
- milieu de résidence
- niveau de vie du ménage
- structure du ménage
- niveau d’éducation de la mère
- âge de la mère à la naissance de l’enfant
- enfin, une variable un peu plus compliquée, à savoir si le rang de naissance de l’enfant (second, troisième, quatrième, etc.) est supérieur au nombre idéal d’enfants selon la mère.
Nous allons préparer les données selon deux approches : soit en utilisant l’extension data.table (voir le chapitre dédié à data.table), soit en utilisant l’extension dplyr (voir le chapitre sur dplyr).
Chargeons les données en mémoire et listons les variables disponibles.
Préparation des données avec data.table
Tout d’abord, regardons sous quel format elles sont stockées.
[1] "tbl_df" "tbl" "data.frame"[1814 obs. x 5 variables] tbl_df tbl data.frame
$id_menage: Identifiant du ménage
numeric: 1 2 3 4 5 6 7 8 9 10 ...
min: 1 - max: 1814 - NAs: 0 (0%) - 1814 unique values
$taille: Taille du ménage (nombre de membres)
numeric: 7 3 6 5 7 6 15 6 5 19 ...
min: 1 - max: 31 - NAs: 0 (0%) - 30 unique values
$sexe_chef: Sexe du chef de ménage
labelled double: 2 1 1 1 1 2 2 2 1 1 ...
min: 1 - max: 2 - NAs: 0 (0%) - 2 unique values
2 value labels: [1] homme [2] femme
$structure: Structure démographique du ménage
labelled double: 4 2 5 4 4 4 5 2 5 5 ...
min: 1 - max: 5 - NAs: 0 (0%) - 5 unique values
6 value labels: [0] pas d'adulte [1] un adulte [2] deux adultes de sexe opposé [3] deux adultes de même sexe [4] trois adultes ou plus avec lien de parenté [5] adultes sans lien de parenté
$richesse: Niveau de vie (quintiles)
labelled double: 1 2 2 1 1 3 2 5 4 3 ...
min: 1 - max: 5 - NAs: 0 (0%) - 5 unique values
5 value labels: [1] très pauvre [2] pauvre [3] moyen [4] riche [5] très richeLes tableaux de données sont au format tibble (c’est-à-dire sont de la classe tbl_df) et les variables catégorielles sont du type haven_labelled (voir le chapitre sur les vecteurs labellisés). Ce format correspond au format de données si on les avait importées depuis SPSS avec l’extension haven (voir le chapitre sur l’import de données).
En premier lieu, il nous faut convertir les tableaux de données au format data.table, ce qui peut se faire avec la fonction setDT. Par ailleurs, nous allons également charger en mémoire l’extension labelled pour la gestion des vecteurs labellisés.
En premier lieu, il nous faut calculer la durée d’observation des enfants, à savoir le temps passé entre la date de naissance (variable du fichier enfants) et la date de passation de l’entretien (fournie par le tableau de données femmes). Pour récupérer des variables du fichier femmes dans le fichier enfants, nous allons procéder à une fusion de table (voir le chapitre dédié). Pour le calcul de la durée d’observation, nous allons utiliser le package lubridate (voir le chapitre calculer un âge et celui sur la gestion des dates). Nous effectuerons l’analyse en mois (puisque l’âge au décès est connu en mois). Dès lors, la durée d’observation sera calculée en mois.
enfants <- merge(
enfants,
femmes[, .(id_femme, date_entretien)],
by = "id_femme",
all.x = TRUE
)
# duree observation en mois
library(lubridate, quietly = TRUE)
enfants[, duree_observation := time_length(interval(date_naissance, date_entretien), unit = "months")]ATTENTION : il y 11 enfants soi-disant nés après la date d’enquête ! Quelle que soit l’enquête, il est rare de ne pas observer d’incohérences. Dans le cas présent, il est fort possible que la date d’entretien puisse parfois être erronnée (par exemple si l’enquêteur a inscrit une date sur le questionnaire papier le jour du recensement du ménage mais n’ai pu effectué le questionnaire individuel que plus tard). Nous décidons ici de procéder à une correction en ajoutant un mois aux dates d’entretien problématiques. D’autres approches auraient pu être envisagées, comme par exemple exclure ces observations problématiques. Cependant, cela aurait impacté le calcul du range de naissance pour les autres enfants issus de la même mère. Quoiqu’il en soit, il n’y a pas de réponse unique. À vous de vous adapter au contexte particulier de votre analyse.
enfants[duree_observation < 0, date_entretien := date_entretien %m+% months(1)]
enfants[, duree_observation := time_length(interval(date_naissance, date_entretien), unit = "months")]Regardons maintenant comment les âges au décès ont été collectés.
Les âges au décès sont ici exprimés en mois révolus. Les décès à un mois révolu correspondent à des décès entre 1 et 2 mois exacts. Par ailleurs, les durées d’observation que nous avons calculées avec time_length sont des durées exactes, c’est-à-dire avec la partie décimale. Pour une analyse de survie, on ne peut mélanger des durées exactes et des durées révolues. Trois approches peuvent être envisagées :
- faire l’analyse en mois révolus, auquel cas on ne gardera que la partie entière des durées d’observations avec la fonction
trunc; - considérer qu’un âge au décès de 3 mois révolus correspond en moyenne à 3,5 mois exacts et donc ajouter 0,5 à tous les âges révolus ;
- imputer un âge au décès exact en distribuant aléatoirement les décès à 3 mois révolus entre 3 et 4 mois exacts, autrement dit en ajoutant aléatoirement une partie décimale aux âges révolus.
Nous allons ici adopter la troisième approche en considérant que les décès se répartissent de manière uniforme au sein d’un même mois. Nous aurons donc recours à la fonction runif qui permets de générer des valeurs aléatoires entre 0 et 1 selon une distribustion uniforme.
Pour définir notre objet de survie, il nous faudra deux variables. Une première, temporelle, indiquant la durée à laquelle survient l’évènement étudié (ici le décès) pour ceux ayant vécu l’évènement et la durée d’observation pour ceux n’ayant pas vécu l’évènement (censure à droite). Par ailleurs, une seconde variable indiquant si les individus ont vécu l’évènement (0 pour non, 1 pour oui). Or, ici, la variable survie est codée 0 pour les décès et 1 pour ceux ayant survécu. Pour plus de détails, voir l’aide de la fonction Surv.
enfants[, deces := 0]
enfants[survie == 0, deces := 1]
var_label(enfants$deces) <- "Est décédé ?"
val_labels(enfants$deces) <- c(non = 0, oui = 1)
enfants[, time := duree_observation]
enfants[deces == 1, time := age_deces_impute]Occupons-nous maintenant des variables explicatives que nous allons inclure dans l’analyse. Tout d’abord, ajoutons à la table enfants les variables nécessaires des tables femmes et menages. Notons qu’il nous faudra importer id_menage de la table femmes pour pouvoir fusionner ensuite la table enfants avec la table menages. Par ailleurs, pour éviter une confusion sur la variable date_naissance, nous renommons à la volée cette variable de la table femmes en date_naissance_mere.
enfants <- merge(
enfants,
femmes[, .(
id_femme, id_menage, milieu, educ,
date_naissance_mere = date_naissance, nb_enf_ideal
)],
by = "id_femme",
all.x = TRUE
)
enfants <- merge(
enfants,
menages[, .(id_menage, structure, richesse)],
by = "id_menage",
all.x = TRUE
)Les variables catégorielles sont pour l’heure sous formes de vecteurs labellisés. Or, dans un modèle, il est impératif de les convertir en facteurs pour qu’elles soient bien traitées comme des variables catégorielles (autrement elles seraient traitées comme des variables continues). On aura donc recours à la fonction to_factor de l’extension labelled.
Regardons plus attentivement, la variable structure.
Tout d’abord, la modalité pas d’adulte
n’est pas représentée dans l’échantillon. On aura donc recours à l’argument drop_unused_labels pour ne pas conserver cette modalité. Par ailleurs, nous considérons que la situation familiale à partir de laquelle nous voudrons comparer les autres dans notre modèle, donc celle qui doit être considérée comme la modalité de référence, est celle du ménage nucléaire. Cette modalité (deux adultes de sexe opposé
) n’étant pas la première, nous aurons recours à la fonction relevel.
enfants[, structure := to_factor(structure, drop_unused_labels = TRUE)]
enfants[, structure := relevel(structure, "deux adultes de sexe opposé")]Regardons la variable educ.
La modalité supérieur
est peu représentée dans notre échantillon. Nous allons la fusionner avec la modalité secondaire
(voir la section Regrouper les modalités d’une variable du chapitre Recodage).
enfants[, educ2 := educ]
enfants[educ == 3, educ2 := 2]
val_label(enfants$educ2, 2) <- "secondaire ou plus"
val_label(enfants$educ2, 3) <- NULL
enfants[, educ2 := to_factor(educ2)]
freq(enfants$educ2)Calculons maintenant l’âge de la mère à la naissance de l’enfant (voir le chapitre Calculer un âge) et découpons le en groupes d’âges (voir la section Découper une variable numérique en classes du chapitre Recodage).
enfants[, age_mere_naissance := time_length(
interval(date_naissance_mere, date_naissance),
unit = "years"
)]
enfants$gpage_mere_naissance <- cut(
enfants$age_mere_naissance,
include.lowest = TRUE, right = FALSE,
breaks=c(13, 20, 30, 50)
)
levels(enfants$gpage_mere_naissance) <- c(
"19 ou moins", "20-29", "30 et plus"
)
enfants$gpage_mere_naissance <- relevel(enfants$gpage_mere_naissance, "20-29")
freq(enfants$gpage_mere_naissance)Reste à calculer si le rang de naissance de l’enfant est supérieur au nombre idéal d’enfants tel que défini par la mère. On aura recours à la fonction rank appliquée par groupe (ici calculé séparément pour chaque mère). L’argument ties.method permet d’indiquer comment gérer les égalités (ici les naissances multiples, e.g. les jumeaux). Comme nous voulons comparer le rang de l’enfant au nombre idéal d’enfants, nous allons retenir la méthode "max" pour obtenir, dans le cas présent, le nombre total d’enfants déjà nés1. Avant de calculer un rang, il est impératif de trier préalablement le tableau (voir le chapitre Tris).
setorder(enfants, id_femme, date_naissance)
enfants[, rang := rank(date_naissance, ties.method = "max"), by = id_femme]
enfants[, rang_apres_ideal := "non"]
# note: unclass() requis en raison d'un bug non corrigé dans haven empéchant de comparer haven_labelled_spss et integer
enfants[rang > unclass(nb_enf_ideal), rang_apres_ideal := "oui"]
enfants[, rang_apres_ideal := factor(rang_apres_ideal)]
enfants[, rang_apres_ideal := relevel(rang_apres_ideal, "non")]Préparation des données avec dplyr
Tout d’abord, regardons sous quel format elles sont stockées.
[1] "tbl_df" "tbl" "data.frame"[1814 obs. x 5 variables] tbl_df tbl data.frame
$id_menage: Identifiant du ménage
numeric: 1 2 3 4 5 6 7 8 9 10 ...
min: 1 - max: 1814 - NAs: 0 (0%) - 1814 unique values
$taille: Taille du ménage (nombre de membres)
numeric: 7 3 6 5 7 6 15 6 5 19 ...
min: 1 - max: 31 - NAs: 0 (0%) - 30 unique values
$sexe_chef: Sexe du chef de ménage
labelled double: 2 1 1 1 1 2 2 2 1 1 ...
min: 1 - max: 2 - NAs: 0 (0%) - 2 unique values
2 value labels: [1] homme [2] femme
$structure: Structure démographique du ménage
labelled double: 4 2 5 4 4 4 5 2 5 5 ...
min: 1 - max: 5 - NAs: 0 (0%) - 5 unique values
6 value labels: [0] pas d'adulte [1] un adulte [2] deux adultes de sexe opposé [3] deux adultes de même sexe [4] trois adultes ou plus avec lien de parenté [5] adultes sans lien de parenté
$richesse: Niveau de vie (quintiles)
labelled double: 1 2 2 1 1 3 2 5 4 3 ...
min: 1 - max: 5 - NAs: 0 (0%) - 5 unique values
5 value labels: [1] très pauvre [2] pauvre [3] moyen [4] riche [5] très richeLes tableaux de données sont déjà au format tibble (c’est-à-dire sont de la classe tbl_df)2 et les variables catégorielles sont du type labelled (voir le chapitre sur les vecteurs labellisés). Ce format correspond au format de données si on les avait importées depuis SPSS avec l’extension haven (voir le chapitre sur l’import de données).
Nous allons charger en mémoire l’extension labelled pour la gestion des vecteurs labellisés en plus de dplyr.
En premier lieu, il nous faut calculer la durée d’observation des enfants, à savoir le temps passé entre la date de naissance (variable du fichier enfants) et la date de passation de l’entretien (fournie par le tableau de données femmes). Pour récupérer des variables du fichier femmes dans le fichier enfants, nous allons procéder à une fusion de table (voir le chapitre dédié). Pour le calcul de la durée d’observation, nous allons utiliser le package lubridate (voir le chapitre calculer un âge et celui sur la gestion des dates). Nous effectuerons l’analyse en mois (puisque l’âge au décès est connu en mois). Dès lors, la durée d’observation sera calculée en mois.
ATTENTION : les étiquettes de valeurs sont le plus souvent perdus lors des fusions de tables avec dplyr. Pour les récupérer après fusion, nous allons conserver une version originale du tableau de données enfants et utiliser la fonction copy_labels_from de l’extension labelled.
enfants_original <- enfants
library(lubridate)
enfants <- enfants %>%
left_join(
femmes %>% dplyr::select(id_femme, date_entretien),
by = "id_femme"
) %>%
copy_labels_from(enfants_original) %>%
mutate(duree_observation = time_length(
interval(date_naissance, date_entretien),
unit = "months"
))ATTENTION : il y 11 enfants soi-disant nés après la date d’enquête ! Quelle que soit l’enquête, il est rare de ne pas observer d’incohérences. Dans le cas présent, il est fort possible que la date d’entretien puisse parfois être erronnée (par exemple si l’enquêteur a inscrit une date sur le questionnaire papier le jour du recensement du ménage mais n’ai pu effectué le questionnaire individuel que plus tard). Nous décidons ici de procéder à une correction en ajoutant un mois aux dates d’entretien problématiques. D’autres approches auraient pu être envisagées, comme par exemple exclure ces observations problématiques. Cependant, cela aurait impacté le calcul du range de naissance pour les autres enfants issus de la même mère. Quoiqu’il en soit, il n’y a pas de réponse unique. À vous de vous adapter au contexte particulier de votre analyse.
enfants$date_entretien[enfants$duree_observation < 0] <-
enfants$date_entretien[enfants$duree_observation < 0] %m+% months(1)
enfants <- enfants %>%
mutate(duree_observation = time_length(
interval(date_naissance, date_entretien),
unit = "months"
))Regardons maintenant comment les âges au décès ont été collectés.
Les âges au décès sont ici exprimés en mois révolus. Les décès à un mois révolu correspondent à des décès entre 1 et 2 mois exacts. Par ailleurs, les durées d’observation que nous avons calculées avec time_length sont des durées exactes, c’est-à-dire avec la partie décimale. Pour une analyse de survie, on ne peut mélanger des durées exactes et des durées révolues. Trois approches peuvent être envisagées :
- faire l’analyse en mois révolus, auquel cas on ne gardera que la partie entière des durées d’observations avec la fonction
trunc; - considérer qu’un âge au décès de 3 mois révolus correspond en moyenne à 3,5 mois exacts et donc ajouter 0,5 à tous les âges révolus ;
- imputer un âge au décès exact en distribuant aléatoirement les décès à 3 mois révolus entre 3 et 4 mois exacts, autrement dit en ajoutant aléatoirement une partie décimale aux âges révolus.
Nous allons ici adopter la troisième approche en considérant que les décès se répartissent de manière uniforme au sein d’un même mois. Nous aurons donc recours à la fonction runif qui permets de générer des valeurs aléatoires entre 0 et 1 selon une distribustion uniforme.
Pour définir notre objet de survie, il nous faudra deux variables. Une première, temporelle, indiquant la durée à laquelle survient l’évènement étudié (ici le décès) pour ceux ayant vécu l’évènement et la durée d’observation pour ceux n’ayant pas vécu l’évènement (censure à droite). Par ailleurs, une seconde variable indiquant si les individus ont vécu l’évènement (0 pour non, 1 pour oui). Or, ici, la variable survie est codée 0 pour les décès et 1 pour ceux ayant survécu. Pour plus de détails, voir l’aide de la fonction Surv.
enfants <- enfants %>%
mutate(deces = if_else(survie == 0, 1, 0)) %>%
set_variable_labels(deces = "Est décédé ?") %>%
set_value_labels(deces = c(non = 0, oui = 1)) %>%
mutate(time = if_else(deces == 1, age_deces_impute, duree_observation))Occupons-nous maintenant des variables explicatives que nous allons inclure dans l’analyse. Tout d’abord, ajoutons à la table enfants les variables nécessaires des tables femmes et menages. Notons qu’il nous faudra importer id_menage de la table femmes pour pouvoir fusionner ensuite la table enfants avec la table menages. Par ailleurs, pour éviter une confusion sur la variable date_naissance, nous renommons à la volée cette variable de la table femmes en date_naissance_mere.
enfants <- enfants %>%
left_join(
dplyr::select(femmes,
id_femme, id_menage, milieu, educ,
date_naissance_mere = date_naissance, nb_enf_ideal
),
by = "id_femme"
) %>%
left_join(
dplyr::select(menages, id_menage, structure, richesse),
by = "id_menage"
) %>%
copy_labels_from(enfants_original) %>%
copy_labels_from(femmes) %>%
copy_labels_from(menages)Les variables catégorielles sont pour l’heure sous formes de vecteurs labellisés. Or, dans un modèle, il est impératif de les convertir en facteurs pour qu’elles soient bien traitées comme des variables catégorielles (autrement elles seraient traitées comme des variables continues). On aura donc recours à la fonction to_factor de l’extension labelled.
Regardons plus attentivement, la variable structure.
Tout d’abord, la modalité pas d’adulte
n’est pas représentée dans l’échantillon. On aura donc recours à l’argument drop_unused_labels pour ne pas conserver cette modalité. Par ailleurs, nous considérons que la situation familiale à partir de laquelle nous voudrons comparer les autres dans notre modèle, donc celle qui doit être considérée comme la modalité de référence, est celle du ménage nucléaire. Cette modalité (deux adultes de sexe opposé
) n’étant pas la première, nous aurons recours à la fonction relevel{data-pkg = “stats”}.
enfants <- enfants %>%
mutate(structure = relevel(
to_factor(structure, drop_unused_labels = TRUE),
"deux adultes de sexe opposé"
))Regardons la variable educ.
La modalité supérieur
est peu représentée dans notre échantillon. Nous allons la fusionner avec la modalité secondaire
(voir la section Regrouper les modalités d’une variable du chapitre Recodage).
enfants <- enfants %>%
mutate(educ2 = ifelse(educ == 3, 2, educ)) %>%
set_value_labels(educ2 = c(
aucun = 0,
primaire = 1,
"secondaire ou plus" = 2
)) %>%
mutate(educ2 = to_factor(educ2))
freq(enfants$educ2)Calculons maintenant l’âge de la mère à la naissance de l’enfant (voir le chapitre Caluler un âge) et découpons le en groupes d’âges (voir la section Découper une variable numérique en classes du chapitre Recodage).
enfants <- enfants %>%
mutate(
age_mere_naissance = time_length(
interval(date_naissance_mere, date_naissance),
unit = "years"
),
gpage_mere_naissance = cut(
age_mere_naissance,
include.lowest = TRUE, right = FALSE,
breaks=c(13, 20, 30, 50)
)
)
levels(enfants$gpage_mere_naissance) <- c(
"19 ou moins", "20-29", "30 et plus"
)
enfants$gpage_mere_naissance <- relevel(enfants$gpage_mere_naissance, "20-29")
freq(enfants$gpage_mere_naissance)Reste à calculer si le rang de naissance de l’enfant est supérieur au nombre idéal d’enfants tel que défini par la mère. On aura recours à la fonction rank appliquée par groupe (ici calculé séparément pour chaque mère). L’argument ties.method permet d’indiquer comment gérer les égalités (ici les naissances multiples, e.g. les jumeaux). Comme nous voulons comparer le rang de l’enfant au nombre idéal d’enfants, nous allons retenir la méthode "max" pour obtenir, dans le cas présent, le nombre total d’enfants déjà nés3. Avant de calculer un rang, il est impératif de trier préalablement le tableau (voir le chapitre Tris).
Kaplan-Meier
La courbe de survie de Kaplan-Meier s’obtient avec la fonction survfit de l’extension survival.
Call: survfit(formula = Surv(time, deces) ~ 1, data = enfants)
n events median 0.95LCL 0.95UCL
[1,] 1584 142 NA NA NAPour la représenter, on pourra avoir recours à la fonction ggsurvplot de l’extension survminer.
Attachement du package : 'survminer'L'objet suivant est masqué depuis 'package:survival':
myelomaWarning: Using `size` aesthetic for lines was deprecated in ggplot2
3.4.0.
ℹ Please use `linewidth` instead.
ℹ The deprecated feature was likely used in the ggpubr
package.
Please report the issue at
<https://github.com/kassambara/ggpubr/issues>.
This warning is displayed once every 8 hours.
Call `lifecycle::last_lifecycle_warnings()` to see where
this warning was generated.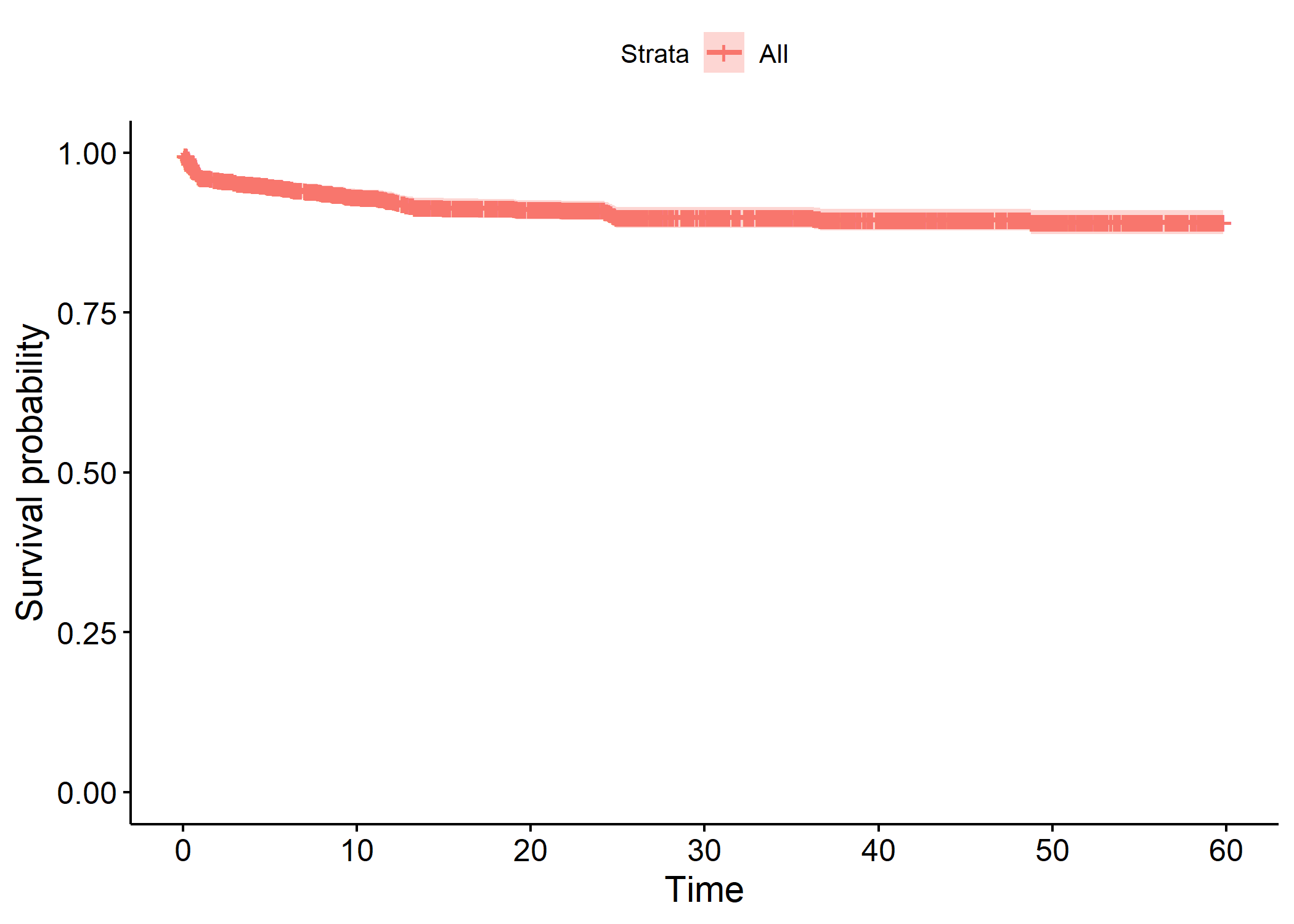
On peut facilement représenter à la place la courbe cumulée des évènements (l’inverse de la courbe de survie) et la table des effectifs en fonction du temps.
Ignoring unknown labels:
• colour : "Strata"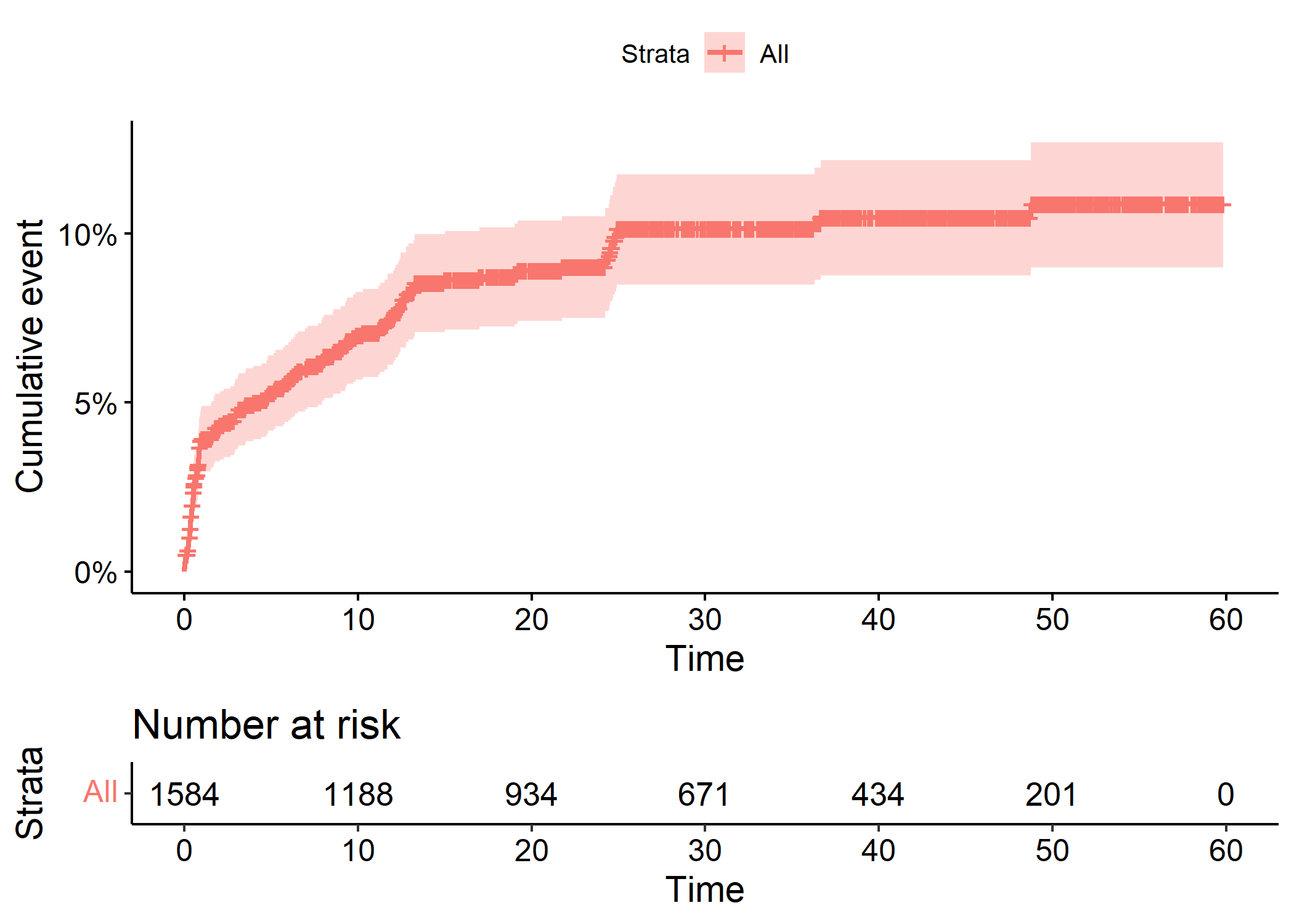
Pour comparer deux groupes (ici les filles et les garçons), il suffit d’indiquer la variable de comparaison à survfit.
Call: survfit(formula = Surv(time, deces) ~ sexe, data = enfants)
n events median 0.95LCL 0.95UCL
sexe=masculin 762 94 NA NA NA
sexe=féminin 822 48 NA NA NALa fonction survdiff permets de calculer le test du logrank afin de comparer des courbes de survie. La mortalité infanto-juvénile diffère-t-elle significativement selon le sexe de l’enfant ?
Call:
survdiff(formula = Surv(time, deces) ~ sexe, data = enfants)
N Observed Expected (O-E)^2/E (O-E)^2/V
sexe=masculin 762 94 66.2 11.6 21.8
sexe=féminin 822 48 75.8 10.2 21.8
Chisq= 21.8 on 1 degrees of freedom, p= 3e-06 Une fois encore, on aura recours à ggsurvplot pour représenter les courbes de survie.
Ignoring unknown labels:
• colour : "Strata"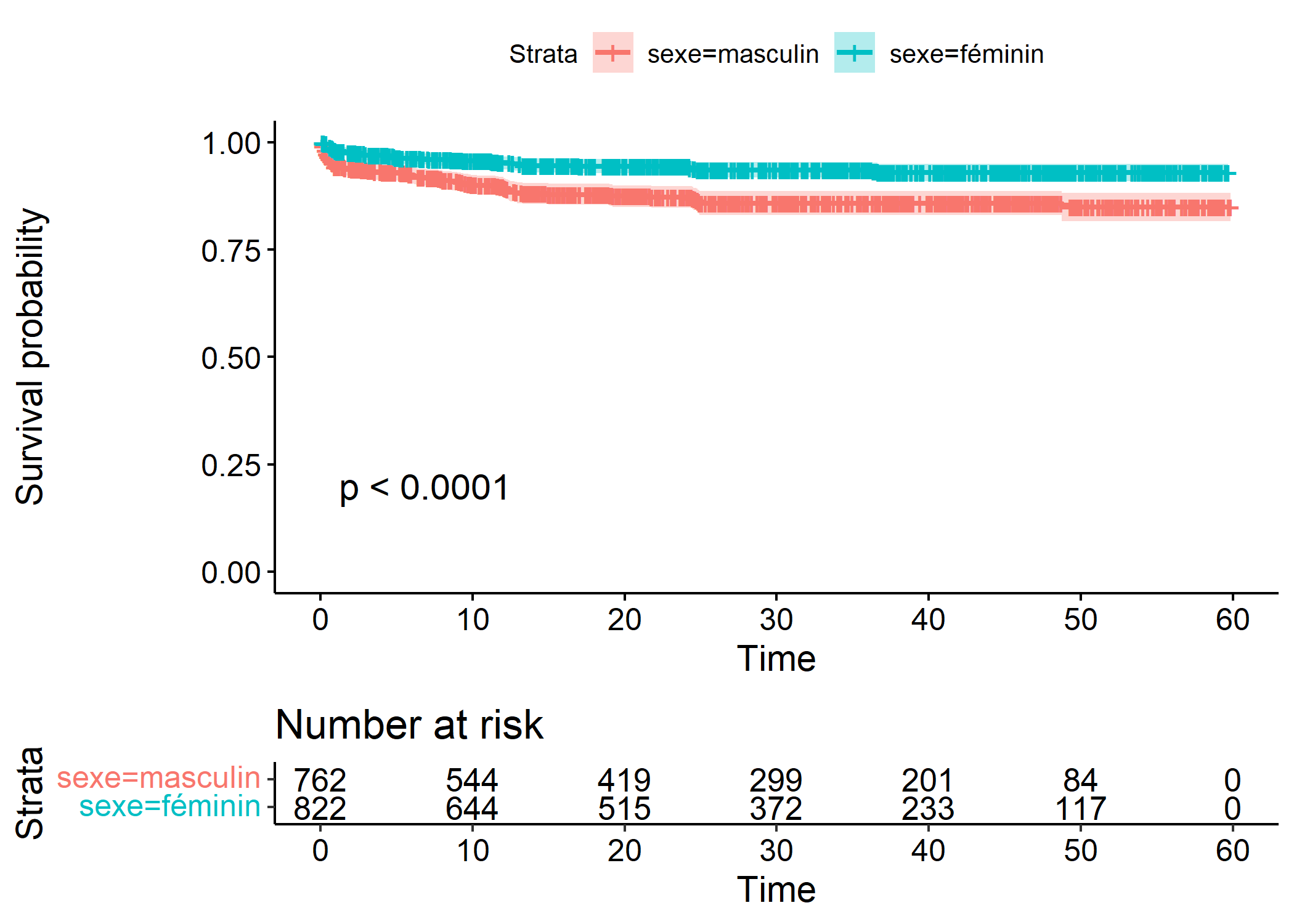
Modèle de Cox
Un modèle de Cox se calcule aisément avec coxph{survival}.
mod1 <- coxph(
Surv(time, deces) ~ sexe + milieu + richesse +
structure + educ2 + gpage_mere_naissance + rang_apres_ideal,
data = enfants
)
mod1Call:
coxph(formula = Surv(time, deces) ~ sexe + milieu + richesse +
structure + educ2 + gpage_mere_naissance + rang_apres_ideal,
data = enfants)
coef
sexeféminin -0.810063
milieu 0.655551
richessepauvre -0.074662
richessemoyen 0.323801
richesseriche 0.349121
richessetrès riche 0.469852
structureun adulte -0.146943
structuredeux adultes de même sexe 0.602041
structuretrois adultes ou plus avec lien de parenté 0.050001
structureadultes sans lien de parenté -0.130140
educ2primaire -0.032561
educ2secondaire ou plus -0.200510
gpage_mere_naissance19 ou moins -0.313232
gpage_mere_naissance30 et plus -0.003499
rang_apres_idealoui 1.364353
exp(coef)
sexeféminin 0.444830
milieu 1.926204
richessepauvre 0.928057
richessemoyen 1.382373
richesseriche 1.417821
richessetrès riche 1.599757
structureun adulte 0.863344
structuredeux adultes de même sexe 1.825841
structuretrois adultes ou plus avec lien de parenté 1.051272
structureadultes sans lien de parenté 0.877972
educ2primaire 0.967963
educ2secondaire ou plus 0.818313
gpage_mere_naissance19 ou moins 0.731080
gpage_mere_naissance30 et plus 0.996507
rang_apres_idealoui 3.913189
se(coef)
sexeféminin 0.177813
milieu 0.269761
richessepauvre 0.250403
richessemoyen 0.247883
richesseriche 0.298204
richessetrès riche 0.428427
structureun adulte 0.600298
structuredeux adultes de même sexe 0.376437
structuretrois adultes ou plus avec lien de parenté 0.196599
structureadultes sans lien de parenté 0.305410
educ2primaire 0.205754
educ2secondaire ou plus 0.366887
gpage_mere_naissance19 ou moins 0.268079
gpage_mere_naissance30 et plus 0.191607
rang_apres_idealoui 0.602554
z
sexeféminin -4.556
milieu 2.430
richessepauvre -0.298
richessemoyen 1.306
richesseriche 1.171
richessetrès riche 1.097
structureun adulte -0.245
structuredeux adultes de même sexe 1.599
structuretrois adultes ou plus avec lien de parenté 0.254
structureadultes sans lien de parenté -0.426
educ2primaire -0.158
educ2secondaire ou plus -0.547
gpage_mere_naissance19 ou moins -1.168
gpage_mere_naissance30 et plus -0.018
rang_apres_idealoui 2.264
p
sexeféminin 5.22e-06
milieu 0.0151
richessepauvre 0.7656
richessemoyen 0.1915
richesseriche 0.2417
richessetrès riche 0.2728
structureun adulte 0.8066
structuredeux adultes de même sexe 0.1098
structuretrois adultes ou plus avec lien de parenté 0.7992
structureadultes sans lien de parenté 0.6700
educ2primaire 0.8743
educ2secondaire ou plus 0.5847
gpage_mere_naissance19 ou moins 0.2426
gpage_mere_naissance30 et plus 0.9854
rang_apres_idealoui 0.0236
Likelihood ratio test=38.16 on 15 df, p=0.000853
n= 1584, number of events= 142 De nombreuses variables ne sont pas significatives. Voyons si nous pouvons, avec la fonction step, améliorer notre modèle par minimisation de l’AIC ou Akaike Information Criterion (voir la section Sélection de modèles du chapitre sur la Régression logistique).
Start: AIC=2027.04
Surv(time, deces) ~ sexe + milieu + richesse + structure + educ2 +
gpage_mere_naissance + rang_apres_ideal
Df AIC
- structure 4 2022.0
- richesse 4 2022.6
- educ2 2 2023.4
- gpage_mere_naissance 2 2024.6
<none> 2027.0
- rang_apres_ideal 1 2028.6
- milieu 1 2031.3
- sexe 1 2047.1
Step: AIC=2021.95
Surv(time, deces) ~ sexe + milieu + richesse + educ2 + gpage_mere_naissance +
rang_apres_ideal
Df AIC
- richesse 4 2016.9
- educ2 2 2018.2
- gpage_mere_naissance 2 2019.4
<none> 2022.0
- rang_apres_ideal 1 2023.4
- milieu 1 2025.5
- sexe 1 2042.2
Step: AIC=2016.91
Surv(time, deces) ~ sexe + milieu + educ2 + gpage_mere_naissance +
rang_apres_ideal
Df AIC
- educ2 2 2013.2
- gpage_mere_naissance 2 2014.8
<none> 2016.9
- rang_apres_ideal 1 2018.0
- milieu 1 2018.7
- sexe 1 2037.5
Step: AIC=2013.18
Surv(time, deces) ~ sexe + milieu + gpage_mere_naissance + rang_apres_ideal
Df AIC
- gpage_mere_naissance 2 2011.1
<none> 2013.2
- rang_apres_ideal 1 2014.2
- milieu 1 2015.6
- sexe 1 2033.7
Step: AIC=2011.1
Surv(time, deces) ~ sexe + milieu + rang_apres_ideal
Df AIC
<none> 2011.1
- rang_apres_ideal 1 2012.3
- milieu 1 2013.9
- sexe 1 2031.5On peut obtenir facilement les coefficients du modèle avec l’excellente fonction tidy de l’extension broom. Ne pas oublier de préciser exponentiate = TRUE. En effet, dans le cas d’un modèle de Cox, l’exponentiel des coefficients corresponds au ratio des risques instantannés ou hazard ratio (HR) en anglais.
Pour représenter ces rapports de risque, on peut ici encore avoir recours à la fonction ggcoef_model de l’extension GGally.
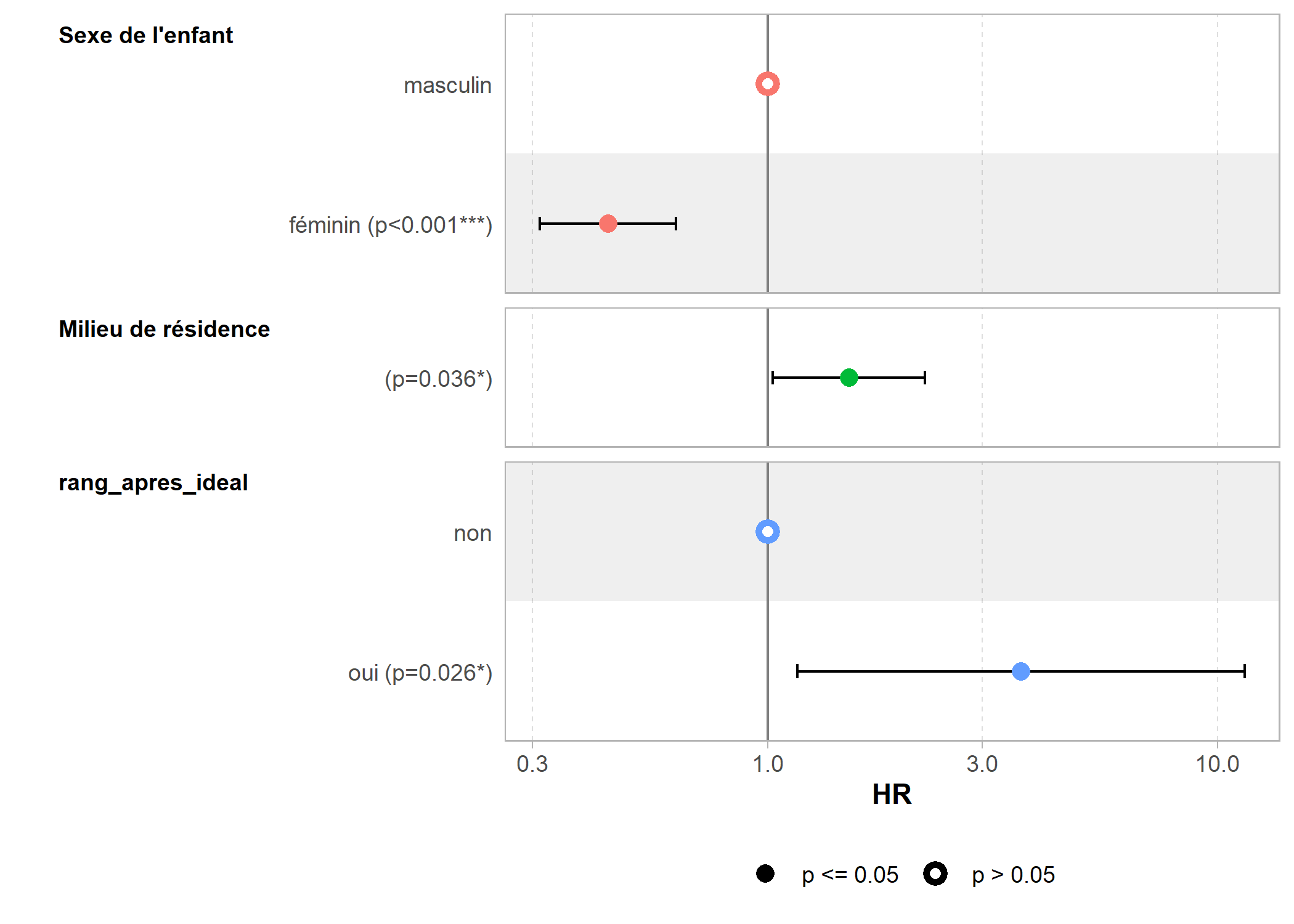
L’extension survminer fournit également une fonction ggforest qui permet de représenter de manière plus esthétique et complète les coefficients d’un modèle de Cox.
Vérification de la validité du modèle
Un modèle de Cox n’est valable que sous l’hypothèse de la proportionnalité des risques relatifs. Selon cette hypothèse les résidus de Schoenfeld ne dépendent pas du temps. Cette hypothèse peut être testée avec la fonction cox.zph.
chisq df p
sexe 0.564 1 0.45
milieu 0.218 1 0.64
rang_apres_ideal 0.226 1 0.63
GLOBAL 1.009 3 0.80Une valeur de p inférieure à 5 % indique que l’hypothèse n’est pas vérifiée. Il apparaît que p est supérieur à 5 % globalement et pour chaque variable prise individuellement. Notre modèle est donc valide.
Il est possible de représenter la distribution des résidus de Schoenfeld à l’aide de ggcoxzph de l’extension survminer, afin de voir si leur répartition change au cours du temps.
Warning in regularize.values(x, y, ties, missing(ties),
na.rm = na.rm): suppression des ex-aequos de 'x'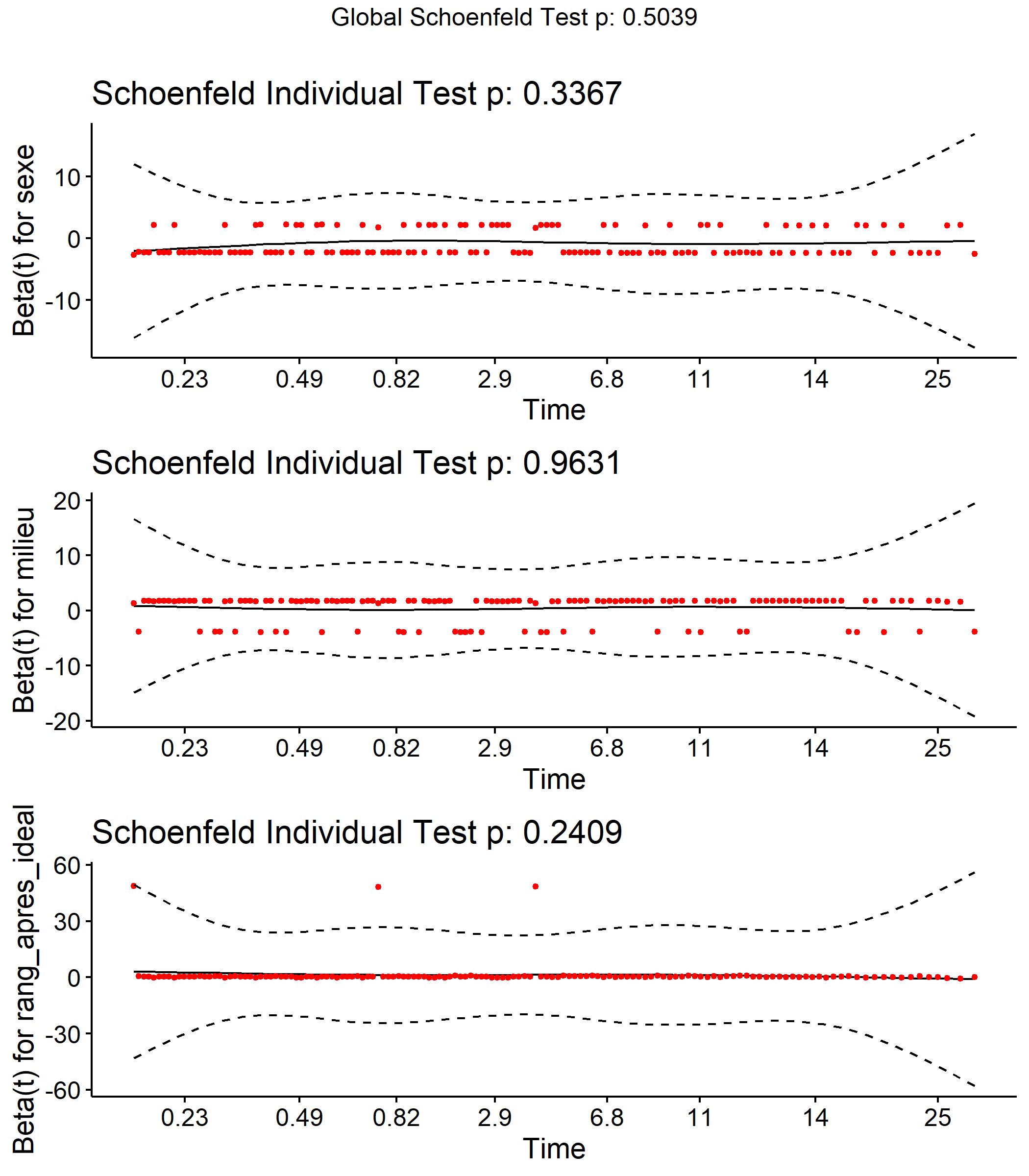
Données pondérées
Si vous utilisez des données pondérées avec un plan d’échantillonnage complexe (voir le chapitre dédié), vous pouvez utilisez les fonctions suivantes de l’extension survey :
svykmpour estimer une courbe de survie de Kaplan-Meier ;svycoxphpour un modèle de Cox.
Dans les deux cas, pensez à ajouter l’option se = TRUE pour que les erreurs standards soient calculées (et que les intervalles de confiance puissent être générés).
Ici, pour plus de simplicité, nous n’avons pas pris en compte les décès éventuels des enfants de rang inférieur avant la naissance considérée.↩︎
Si cela n’avait pas été le cas, nous aurions eu recours à la fonction
tbl_df.↩︎Ici, pour plus de simplicité, nous n’avons pas pris en compte les décès éventuels des enfants de rang inférieur avant la naissance considérée.↩︎

